Краткая информация о проекте
(2023-2025 г.г.)
Наименование проекта: ИРН AP19678215 «Метагеномный и популяционный анализ вирусов и вироидов картофеля и изучение механизмов их взаимодействия с устойчивыми сортами и гибридами картофеля».
Актуальность.
Данный проект нацелен на решение проблем, связанных с контролем над распространением вирусных инфекций картофеля, предотвращением завоза опасных штаммов и изолятов экономически значимых вирусов и вироидов из других стран, с качественным повышением селекционного потенциала сортов, устойчивых к вирусной инфекции. Интенсификация картофелеводства в стране путем культивирования устойчивых локальных сортов картофеля и контроль над распространением вирусов и их переносчиков позволит прийти к импортозамещению и зависимости от зарубежного семенного материала. Молекулярно-генетическое изучение вирусов картофеля и их взаимодействие с сортами картофеля локальной селекции позволит предотвратить распространение вирусов на картофельных полях, особенно семеноводческих хозяйств.
Цель проекта:
Целью проекта является исследование вирома картофеля, изучение генетического разнообразия вирусов и вироидов картофеля, распространенных на территории Казахстана, а также механизмов взаимодействия природных изолятов вирусов и вироидов с устойчивыми сортами картофеля и выявление новых генов, ассоциированных с иммунным ответом на вирусную инфекцию.
Ожидаемые результаты:
1) Будет проведен сбор не менее 500 образцов картофеля в картофелеводческих регионах страны (Северный регион и Алматинская область). Метагеномный анализ вирома картофеля, видовая идентификация вирусов и вироидов, распространенных на картофелеводческих полях страны. Обнаружение новых, карантинных и особо опасных вирусов и вироидов. Для отбора будет производиться сбор клубней и надземной части у растений с признаками поражения, а также у бессимптомных растений. Клубни будут использоваться как источник вирионов для инокуляции устойчивых растений.
2) Будет проведено полногеномное секвенирование обнаруженных PLRV, X, Y, S и M вирусов и PSTVd вироида картофеля, приносящих наибольший экономический урон, обнаруженние изолятов и штаммов, циркулирующих в стране. Разработка нового метода vir-типирования вирусов для определения генетического разнообразия изолятов в географически отдаленных популяциях. Сравнение результатов популяционного анализа при vir-типировании и полногеномном секвенировании. Новый метод типирования позволит в разы снизить затраты на изучение популяционной генетики вирусов. Определение природных штаммов/изолятов со значимыми мутациями в белках, ответственных за амплификацию, передвижение и супрессию РНК-интерференции, выявление влияния мутаций на биологию вируса методом инокуляции. Выявление уровня внутривидовой и межвидовой рекомбинации. Выявление новых рекомбинантных форм. Полногеномное секвенирование позволит выявить консервативные и вариабельные паттерны, влияющие на инфекционные способности вируса.
3) Будет проведено выявление интрогрессных генов устойчивости к экономически важным вирусам и вироидам картофеля у 250 сортов и гибридов картофеля Solanum tuberosum местной и зарубежной селекции. В селекции локальных сортов и гибридов картофеля задействованы разнообразные исходные линии, что приводит к положительной вероятности определения устойчивых аллелей, как это было показано нами при исследовании данных сортов на наличие аллелей устойчивости к X и A вирусам картофеля.
4) Будет проведена инокуляция отобранных сортов картофеля разными комбинациями мутантов в генах, кодирующих белки, ответственные за амплификацию, передвижение и супрессию РНК-интерференции. Определение уровня амплификации и системного передвижения разных изолятов вируса в растениях, несущих гены устойчивости. Определение взаимосвязи активности генов устойчивости в определенной генетической среде (разные сорта) к отобранным изолятам вируса. Выявление влияния генетической среды на активность генов устойчивости. Выявление патогенности отобранных изолятов/штаммов.
5) Будет проведен транскриптомный анализ сортов картофеля, инокулированных изолятами вирусных патогенов, несущих значимые мутации в целевых генах, выявление новых генов, участвующих в устойчивости картофеля к вирусным патогенам, в том числе при инокуляции несколькими вирусами.
6) Будет создан банк образцов новых вирусных патогенов, штаммов и изолятов. Разработка методик для селекционеров с описанием генетических паспортов по генам устойчивости к экономически значимым вирусам и степени устойчивости для каждого сорта.
Научный руководитель проекта:
Гриценко Диляра Александровна
Члены исследовательской группы:
Гриценко Д.А. – заведующая лабораторией
Низамдинова Г, К. — СНС
Пожарский А.С.- НС
Таскужина А.К.- МНС
Капытина А.И.- МНС
Керімбек Н.М- МНС
Костюкова В.С.- МНС
Колченко М.В. — лаборант
Абдрахманова А. Б.- лаборант
Адильбаева К.- лаборант
Махамбетов А.Н.- лаборант
Список публикаций и патентов исполнителей проекта за период 2018-2022 гг.
- Gritsenko, D., et al. Development of a “deconstructed” vector based on the genome of grapevine virus A // Plant Biotechnol Rep. -2019. Индекс цитирования – 2, Процентиль – 64, Квартиль- Q2, DOI: 10.1007/s11816-019-00528-1.
- Pozharskiy, A., Kostyukova, V., Nizamdinova, G., Kalendar, R., & Gritsenko, D. (2022). MLO proteins from tomato (Solanum lycopersicum L.) and related species in the broad phylogenetic context. Plants, 11(12), 1588.. DOI: 10.3390/plants11121588; WOS. Q1 (IF 4,827), Scopus: процентиль 71.
- Gritsenko, D., Pozharskiy, A., Dolgikh, S., Aubakirova, K., Kenzhebekova, R., Galiakparov, N., Sadykov, S. Apple varieties from Kazakhstan and their relation to foreign cultivars assessed with RosBREED 10K SNP array. 2022. Eur.J.Hortic.Sci. 87 (1) 1-8, DOI: 10.17660/eJHS.2022/006. Индекс цитирования – 0, Процентиль – 62, Квартиль- Q2.
- Pozharskiy, A., Kostyukova, V., Taskuzhina, A., Nizamdinova, G., Kisselyova, N., Kalendar, R., Gritsenko, D. (2022). Screening a collection of local and foreign varieties of Solanum lycopersicum L. in Kazakhstan for genetic markers of resistance against three tomato viruses. Heliyon. DOI: 10.1016/j.heliyon.2022.e10095; WOS. Q2 (IF-3.7), Scopus: процентиль 82.
- Gritsenko, D., Zulfiya Kachiyeva, Gulzhan Zhamanbayev, Bakhytzhan DuisembekoV, Abai Sagitov. Detection of five potato viruses in Kazakhstan // IX International scientific agriculture symposium “AGROSYM 2018”., p. 611. 2018.
- Gritsenko D., Aubakirova K., Galiakrapov N. Simultaneous detection of five apple viruses by RT-PCR. International Journal of Biology and Chemistry (2020) v. 13, n. 1, p. 129-134. 2020. Индекс цитирования –0, doi: 10.26577/ijbch.2020.v13.i1.13.
- 7. Gritsenko, D., et al. «Detection of Grapevine virus A in wild grape in Kazakhstan.» Phytopathology. Vol. 109. No. 11. 3340 Pilot Knob Road, St Paul, Mn 55121 Usa: Amer Phytopathological Soc, 2019.
- Gritsenko, D. A., K. P. Aubakirova, and A. S. Pozharskiy. «SSR profiling of potato cultivars resistant to pathogens.» Plant Genetics, Genomics, Bioinformatics, and Biotechnology. 2021.
- A.S. Pozharskiy, K. Aubakirova, D. Gritsenko, N. Galiakparov. Genotyping and morphometric analysis of Kazakhstani grapevine cultivars versus Asian and European cultivars // Genet. Mol. Res., 2020. Индекс цитирования – 0, DOI: 10.4238/gmr18482.
- A. Pozharskiy, D. Gritsenko // Prediction of Slmlo1 protein paralogs in Solanum l. Spp. using partially assembled genomic dat // IV. International Agricultural, Biological & Life Science Conference – 2022 – P.162.
- A. Kapytina, N. Kerimbek, A. Taskuzhina, G. Nizamdinova, K. Adilbayeva, S. Murzatayeva, Z. Kachiyeva // Detection and genetic investigation of potato leafroll virus in Kazakhstan // IV. International Agricultural, Biological & Life Science Conference – 2022 – P.165.
- Керимбек Н., Капытина А., Пожарский А., Хуснитдинова М., Гриценко Д. // Филогенетический анализ вируса кустистой карликовости малины // Сборник материалов Международной научно-практической конференции «Актуальные проблемы и перспективы развития науки в области плодоовощеводства» -2022 – С.139
Достигнутые результаты за 2023 г.:
Был проведен анализ нуклеотидных последовательностей полного генома или отдельных регионов для вируса PLRV в количестве 296, для вируса PVX- 720, PVY – 632, PVS – 439, PVM – 276, PSTVd – 2864. В результате анализа было выявлено не менее 5 перспективных регионов для дизайна праймеров для каждого целевого патогена. Вариабельность в пределах геномов составила от 3,4 до 15,3 %, в пределах отдельных генов не превышала в среднем 4,7%. Для дизайна праймеров использовался регион, консервативность которого в разрезе вида составляла не менее 99,5% при анализе разных штаммов и изолятов, вариабельность за пределами вида составляла не менее 60%.
В результате анализа для лабораторной коллекции картофеля были выявлены вирусы PLRV (11%), PVX (37%), PVY (29%) и Alfalfa mosaic virus (4%). Гомология среди изолятов каждого вируса PLRV, PVX и PVY при полногеномном анализе составила 87 — 95%. Среди изолятов PLRV частота мутаций не превышала 1.3 × 10−3 , для PVX 1.01 × 10−3 , для вируса PVY 1.15 × 10−3. Наибольшая частота мутаций была отмечена у вируса PLRV.
Был проведен метагеномный анализ надземной части растений, а также клубней картофеля, собранных с полей. В результате анализа были выявлены вирусы PLRV, X, Y, S, а также вироид PSTVd. В северном регионе страны доминируют вирусы X и Y, в то время как в южном регионе (Нарынколь) доминирует вирус X. Помимо вышеуказанных вирусов, также был выявлен вирус Alfalfa mosaic virus.
Полногеномный анализ вирусов PLRV, PVX и PVY показал высокую гомологию внутри каждой исследуемой популяции изолятов, которая составила 97 — 100%. Среди изолятов PLRV частота мутаций варьировала от 0.95 × 10−3 до 1.19 × 10−3 , для PVX от 0.82 × 10−3 до 0.98 × 10−3 , для вируса PVY не превышала 0.92 × 10−3.
Достигнутые результаты за 2024 год
Было проведено полногеномное секвенирование 43, 48 и 11 изолятов вирусов S, M и вироида PSTVd, с глубиной покрытия генома от 180 до 250Х, обеспечившей полное покрытие генома (100%). В результате Vir-типирования было получено 132 полиморфных ампликона для вируса S, 161 для вируса M и 115 для PSTVd. Вирус M показал высокий уровень вариативности как внутри регионов (Северный и Южный), так и между ними, с генетической дистанцией 0,124, при этом гомология внутри регионов не превышала 89%. Филогенетический анализ показал, что локальные изоляты вируса S образуют субклады с изолятами PVSI, а изоляты вируса M — с изолятами PVM-o. Для вируса Х была установлена генетическая дистанция между популяциями (0,198) и выделение отдельных клад для каждого региона. Локальные изоляты вируса Y сформировали субгруппы с филлогруппой I-1, а генетическая дистанция внутри популяции составила 0,087. Наибольшее родство с изолятами было найдено с филлогруппой O. Для вируса PLRV наибольшую вариабельность продемонстрировали открытые рамки считывания, кодирующие RTD и P0, с уровнями вариабельности 35,62% и 27,615% соответственно, с выявлением 9 значимых мутаций, влияющих на биологическую активность белков. Для вирусов S и M капсидные белки оказались наиболее вариабельными, с уровнями вариабельности 21% и 19,3% соответственно, при этом для вируса M было обнаружено 5 уникальных вариантов, влияющих на биологическую активность белков. Вирус Y показал наибольшую вариабельность в генах, кодирующих белки Р1 и VPg, с уровнями вариабельности 21,3% и 12,1%, но значимых мутаций, влияющих на биологическую активность белков, выявлено не было. Для вируса X наибольшую вариабельность показал ген, кодирующий капсидный белок, с уровнем вариабельности 19,8%, и было выявлено 15 значимых мутаций, влияющих на сборку вирусной частицы и ее передвижение, включая 3 мутации в предполагаемом регионе взаимодействия с геномом. Для исследования было отобрано 50 образцов картофеля. Выборка включала сорта и гибриды с различными агрономическими характеристиками, адаптированные к климатическим условиям Казахстана. Основные сорта включают как раннеспелые, так и позднеспелые сорта с различной урожайностью и полевой устойчивостью к основным вирусным патогенам. Для подтверждения качества геномной ДНК и их пригодности к дальнейшему анализу был проведен качественный контроль путем амплификации гена домашнего хозяйства 28rRNA. Для скрининга генов, отвечающих за устойчивость к вирусам картофеля были отобраны следующие маркеры: для устойчивости к вирусу Y – ADG1, ADG2, RysC3, RY186, GP122/718, GP122/564, SCARysto4; для вируса X – SPUD237, CP60, PVX; для вируса PRLV – RGASC850; и для вируса PVS – SC811/260, CP16. Результаты показали, что сорта Максим, Ресурс, Беркут, а также гибрид Ts-6 имеют наибольшее количество локусов, связанных с устойчивостью к данным вирусным патогенам. Клубни инфицированного картофеля проращивали в контролируемых условиях в климат камере. Для проращивания использовали образцы, которые были ранее протестированы на наличие вирусных патогенов, с каждого региона было отобрано не менее 10 клубней, инфицированных каждым вирусом по-отдельности, а также клубни, зараженные несколькими вирусами одновременно.
Публикации и патенты
l Adilbayeva, K., Moisseyev, R., Kolchenko, M., Kenzhebekova, R., Khassanov, V., Beisembina, B., … & Gritsenko, D. (2024). Genetic Evaluation of Kazakhstani Potato Germplasm for Pathogen and Pest Resistance Using DNA Markers. Agronomy, 14(9), 1923.
2 Кенжебекова, Р. Т., Мендыбаева, А. С., Капытина, А. И., & Гриценко, Д. А. (2024). Распространение смешанных вирусных инфекций картофеля в алматинской области казахстана. Вестник КазНУ. Серия биологическая, 99(2), 91-100.
Полученные результаты по задачам 2025:
Был проведен транскриптомный анализ сортов картофеля, инфицированных вирусами PVY, PVX, PVS, PLRV и PVM на 21 dpi, с целью выявления экспрессии генов, связанных с механизмами устойчивости к вирусным инфекциям. Данный анализ позволил установить закономерности регуляции генов, участвующих в механизмах противовирусной устойчивости, и выявил существенные различия между сортами по уровню транскрипционной активности.
На основе данных высокопроизводительного секвенирования были построены профили экспрессии генов, проведен анализ корреляций между транскрипционными паттернами, а также выполнено функциональное аннотирование наиболее экспрессируемых генов в среднем для всех вирусов и их комбинаций.
Анализ профилей экспрессии позволил выделить восемнадцать наиболее активно транскрибируемых генов у исследованных сортов картофеля. Уровень транскрипционной активности значительно варьировал между генотипами: наибольшие значения экспрессии отмечены у сортов Беркут и Жигулевский, тогда как у сортов Дина и Максим наблюдались минимальные показатели.
Функциональный анализ наиболее экспрессируемых генов показал, что активные транскрипты преимущественно вовлечены в ключевые метаболические и регуляторные процессы, обеспечивающие адаптацию растения к биотическим стрессам, включая вирусную инфекцию.
Проведенный корреляционный анализ показал, что между сортами картофеля наблюдается высокая степень сопряженности экспрессии основных генов, участвующих в противовирусной защите и фотосинтезе.
На основе результатов молекулярной детекции, полногеномного секвенирования и метагеномного анализа создан банк образцов исследованных патогенов, включающий молекулярно охарактеризованные изоляты вирусов PLRV, PVX, PVY, PVS, PVM и вироида PSTVd.
Были опубликованы научно-методические рекомендации, предназначенные для использования в селекции, направленные на молекулярно-генетическую паспортизацию сортов, гибридов и линий картофеля по генам устойчивости к экономически значимым вирусам с оценкой степени устойчивости каждого генотипа.
Информация для потенциальных пользователей
Потенциал применения результатов проекта в научной и производственной среде является значительным. Разработанные тест-системы могут быть внедрены в мониторинг вирусных инфекций картофеля для оперативного выявления угроз и предотвращения их распространения на посевных площадях. Выявленные генотипы картофеля, несущие гены устойчивости к экономически значимым вирусынм патогенам, являются важной базой для направленной селекции высокоурожайных сортов. Полученные научные материалы и методические разработки доступны для использования в высших учебных заведениях Республики Казахстан.
Эффективность проведённых исследований подтверждается выполнением всех этапов проекта и получением высокозначимых научных результатов опубликованных в высокорейтинговых изданиях. Проведённый комплексный генетический анализ изолятов вирусов и вироидов картофеля позволил выявить новые варианты патогенов, характерных для Казахстана, а также определить их связь со степенью устойчивости у различных сортов местной и зарубежной селекции. Были выявлены гены, участвующие в формировании иммуннитета растений к вирусной инфекции, включая случаи смешанных инфекций. Полученные результаты формируют научную основу для разработки инновационных методов селекции и производства оздоровленного посадочного материала без необходимости вмешательства в геном картофеля, что повышает технологическую и экономическую эффективность защиты растений от вирусных заболеваний.
Патенты:
- Гриценко Д.А., Керімбек Н.М., Адильбаева К.С., Дулат Б. Патент на полезную модель № 10787 (2025) «Набор синтетических олигонуклеотидов для обнаружения вируса скручивания листьев картофеля»
Научно-методические рекомендации:
1) Адильбаева К.С., Моисеев Р.М., Капытына А.И., Проценко Е.В., Гриценко Д.А. (2025). Молекулярно-генетическая паспортизация сортов, гибридов и линий картофеля отечественной и зарубежной селекции. — Алматы: Жебе, — 76 с. — ISBN 978-601-08-5440-6.
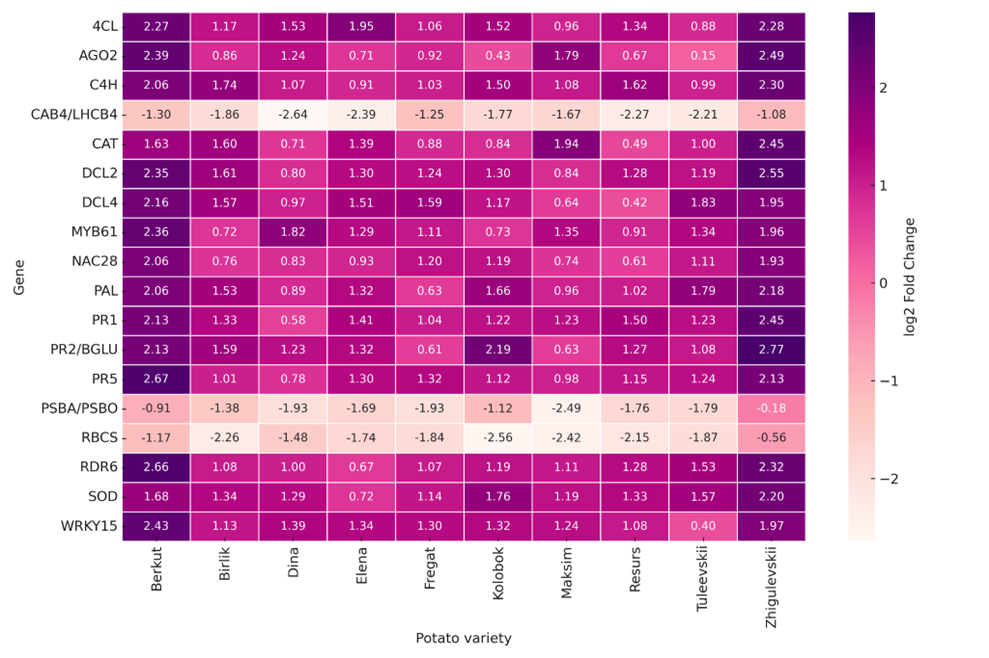
Рисунок 1 – Тепловая карта уровней экспрессии наиболее активных генов у сортов картофеля. Тепловая карта визуализирует нормализованные значения экспрессии (от –3 до +3) для восемнадцати наиболее транскрибируемых генов в различных сортах картофеля. По оси X представлены сорта картофеля, по оси Y — гены с наибольшей экспрессией. Цветовая шкала отражает уровень транскрипционной активности, где отрицательные значения указывают на пониженную, а положительные — на повышенную экспрессию генов относительно среднего уровня.
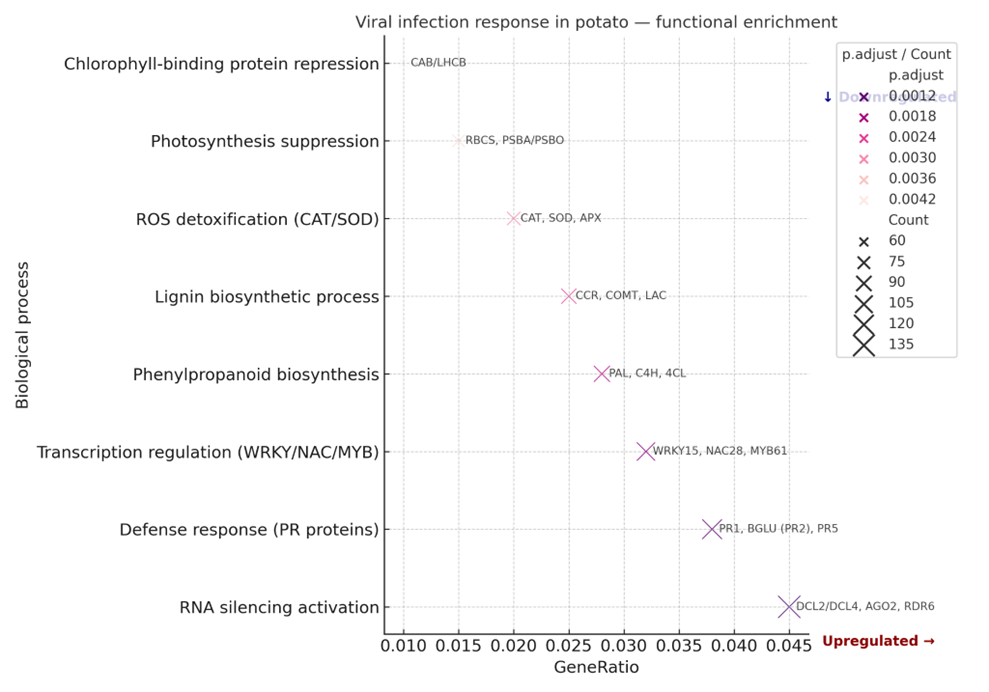
Рисунок 2 – Функциональная аннотация наиболее экспрессируемых генов (GO-анализ). По оси X представлено отношение числа генов, участвующих в процессе (GeneRatio), по оси Y — названия биологических процессов. Цветовая шкала отражает уровень статистической значимости (p.adjust), а размер точек соответствует количеству генов, вовлеченных в соответствующий процесс
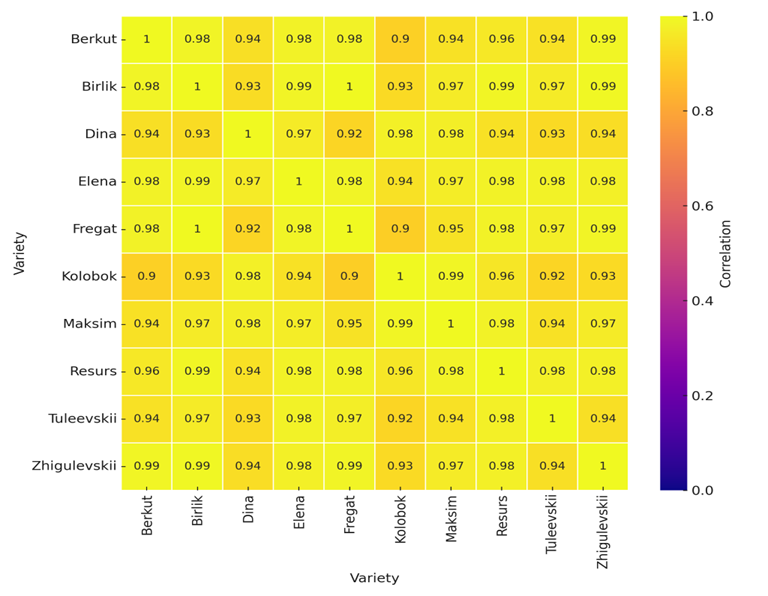
Рисунок 3 – Матрица корреляции экспрессии генов у сортов картофеля. По осям X и Y указаны наименования сортов, а цветовая шкала отображает коэффициенты корреляции Пирсона (от 0 до 1): фиолетовые оттенки соответствуют низкой степени сходства профилей экспрессии, а желтые – высокой корреляции между сортами.

