Краткая информация о проекте
(2023-2025 г.г.)
Наименование проекта: ИРН AP19679755 «Полногеномное и молекулярно-генетическое изучение Erwnina amylovora и грибковых патогенов рода Venturia в диких и культурных популяциях плодовых деревьев».
Актуальность.
Данный проект нацелен на решение проблем, связанных с распространением экономически значимых и карантинных фитопатогенов в стране, предотвращением завоза в страну новых и высокопатогенных штаммов, с распространением инфекции в диких популяциях плодовых деревьев, что приводит к сокращению генетического и биологического разнообразия. Результаты данного проекта позволят выявить новые варианты и гены вирулентности и патогенности возбудителей бактериального ожога и парши, что позволит разработать направленные методы борьбы с ними. Предотвращение распространение инфекции в культурных садах и диких популяций будет осуществлено путем анализа естественных резервуарах патогенов и выявления очагов инф екции в естественных и искусственно созданных условиях. Выявление новых рас и штаммов позволит оценить их уровень патогенности и своевременно ограничить их распространение. В настоящий момент, в стране не определяются риботипы, штаммы Erwnina amylovora, а также расы грибкового патогена рода Venturia, не определяется регион их распространения и применяются одинаковые методы борьбы без полной эрадикации инфекции, что приводит к гибели целых садов. Полногеномные исследования позволят оценить темпы эволюции патогена в стране и предсказать возникновение высокопатогенных штаммов.
Цель проекта:
Целью проекта является полногеномное и молекулярно-генетическое изучение разных штаммов и изолятов Erwnina amylovora, Venturia inaequalis, Venturia pirina и Venturia asperata циркулирующих в диких и культурных популяциях яблони и груши, а также в естественных резервуарах для анализа эволюции и патогенности возбудителей бактериального ожога и парши плодовых культур.
Ожидаемые результаты:
1) Будет проведен сбор не менее 600 образцов растительного материала в популяциях дикой яблони на территории Джунгарского и Заилийского Алатау, а также у культурной яблони и груши в садах южного Казахстана. Сбор растительного материала у растений Рябины и Боярышника, естественных резервуарах патогена Erwnina amylovora. Будут отбираться образцы цветков, веточек и листьев в весеннее и летнее время. Для каждого дерева будут отмечены GPS координаты. Сбор образцов в естественных резервуарах будет производиться у растений, произрастающих в непосредственной близости с популяциями яблони и груши, а также удаленных не менее чем на 1 км от популяций дикой и культурной яблони.
2) Будет проведен метагеномный анализ надземной части растений и определение основных бактериальных и грибковых патогенов, поражающих яблоню, грушу, боярышник и рябину. Выявление новых бактерий и грибков, ранее не изученных в Казахстане, определение сопутствующей микрофлоры и смешанной инфекции. Образцы положительные на наличие Erwnina amylovora и рода Venturia будут анализироваться дополнительно петлевой изотермической амплификацией со специфичными праймерами к геному патогена. Для метагеномного анализа будет проводиться амплификация полного гена 16S РНК бактериальных и ITS регионов грибковых патогенов методом нанопорового секвенирования для повышения видовой разрешающей способности. Будут выявлены образцы со смешанной инфекцией, включающей бактериальный ожог и паршу.
3) Будет проведено полногеномное секвенирование штаммов и изолятов Erwnina amylovora, Venturia inaequalis, Venturia pirina и Venturia asperata, обнаруженных у растений яблони, груши, рябины и боярышника. Будет проведен популяционный и филогенетический анализы с определением уровня гетерогенности и эволюционного вектора патогенов. Выявление высоковариабельных и консервативных регионов геномов, уровень вариабельности всего генома и в зависимости от хозяина и региона распространения. Будут определены значимые мутации в генах вирулентности, выявлены новые варианты. Будут определены перспективные регионы геномов для высокочувствительной детекции патогенов. Будут впервые в стране идентифицированы известные и новые расы рода Venturia и риботипы Erwnina amylovora.
4) Будет проведен анализ экспрессии генов вирулентности патогенов в условиях in vitro и у зараженных образцов яблони и груши, определение транскриптомных профилей с выявлением новых вариантов генов вирулентности и их сочетаний. Будет проведен сравнительный анализ экспрессии генов вирулентности у разных штаммов и изолятов.
5) Будет разработана информационная карта циркуляции Erwnina amylovora и грибковых патогенов рода Venturia в популяциях дикой и культурной яблони и груши в стране.
6) Будут получены чистые культуры патогенов и создана коллекция штаммов, риботипов и изолятов, распространенных в стране. Коллекция патогенов будет являться референтной для анализа эволюции патогенов, идентификации новых рас и штаммов, предсказания вектора эволюции.
Научный руководитель проекта:
Низамдинова Гульназ Карамидиновна
Члены исследовательской группы:
Гриценко Д.А.- зав. лаб
Низамдинова Г, К.- СНС
Пожарский А.С.- НС
Таскужина А.К.- МНС
Капытина А.И.- МНС
Керімбек Н.М- МНС
Костюкова В.С.- МНС
Колченко М.В.- лаборант
Абдрахманова А. Б.- лаборант
Адильбаева К.- лаборант
Список публикаций и патентов исполнителей проекта за период 2018-2022 гг.
- Gritsenko, D., et al. Development of a “deconstructed” vector based on the genome of grapevine virus A // Plant Biotechnol Rep. -2019. Индекс цитирования – 2, Процентиль – 64, Квартиль- Q2, DOI: 10.1007/s11816-019-00528-1.
- Pozharskiy, A., Kostyukova, V., Nizamdinova, G., Kalendar, R., & Gritsenko, D. (2022). MLO proteins from tomato (Solanum lycopersicum L.) and related species in the broad phylogenetic context. Plants, 11(12), 1588.. DOI: 10.3390/plants11121588; WOS. Q1 (IF 4,827), Scopus: процентиль 71.
- Gritsenko, D., Pozharskiy, A., Dolgikh, S., Aubakirova, K., Kenzhebekova, R., Galiakparov, N., Sadykov, S. Apple varieties from Kazakhstan and their relation to foreign cultivars assessed with RosBREED 10K SNP array. 2022. Eur.J.Hortic.Sci. 87 (1) 1-8, DOI: 10.17660/eJHS.2022/006. Индекс цитирования – 0, Процентиль – 62, Квартиль- Q2.
- Pozharskiy, A., Kostyukova, V., Taskuzhina, A., Nizamdinova, G., Kisselyova, N., Kalendar, R., Gritsenko, D. (2022). Screening a collection of local and foreign varieties of Solanum lycopersicum L. in Kazakhstan for genetic markers of resistance against three tomato viruses. Heliyon. DOI: 10.1016/j.heliyon.2022.e10095; WOS. Q2 (IF-3.7), Scopus: процентиль 82.
- Gritsenko, D., Zulfiya Kachiyeva, Gulzhan Zhamanbayev, Bakhytzhan DuisembekoV, Abai Sagitov. Detection of five potato viruses in Kazakhstan // IX International scientific agriculture symposium “AGROSYM 2018”., p. 611. 2018.
- Gritsenko D., Aubakirova K., Galiakrapov N. Simultaneous detection of five apple viruses by RT-PCR. International Journal of Biology and Chemistry (2020) v. 13, n. 1, p. 129-134. 2020. Индекс цитирования –0, doi: 10.26577/ijbch.2020.v13.i1.13.
- Gritsenko, D., et al. «Detection of Grapevine virus A in wild grape in Kazakhstan.» Phytopathology. Vol. 109. No. 11. 3340 Pilot Knob Road, St Paul, Mn 55121 Usa: Amer Phytopathological Soc, 2019.
- Gritsenko, D. A., K. P. Aubakirova, and A. S. Pozharskiy. «SSR profiling of potato cultivars resistant to pathogens.» Plant Genetics, Genomics, Bioinformatics, and Biotechnology. 2021.
- A.S. Pozharskiy, K. Aubakirova, D. Gritsenko, N. Galiakparov. Genotyping and morphometric analysis of Kazakhstani grapevine cultivars versus Asian and European cultivars // Genet. Mol. Res., 2020. Индекс цитирования – 0, DOI: 10.4238/gmr18482.
- A. Pozharskiy, D. Gritsenko // Prediction of Slmlo1 protein paralogs in Solanum l. Spp. using partially assembled genomic dat // IV. International Agricultural, Biological & Life Science Conference – 2022 – P.162.
- A. Kapytina, N. Kerimbek, A. Taskuzhina, G. Nizamdinova, K. Adilbayeva, S. Murzatayeva, Z. Kachiyeva // Detection and genetic investigation of potato leafroll virus in Kazakhstan // IV. International Agricultural, Biological & Life Science Conference – 2022 – P.165.
- Керимбек Н., Капытина А., Пожарский А., Хуснитдинова М., Гриценко Д. // Филогенетический анализ вируса кустистой карликовости малины // Сборник материалов Международной научно-практической конференции «Актуальные проблемы и перспективы развития науки в области плодоовощеводства» -2022 – С.139
Достигнутые результаты за 2023 г.:
Был проведен сбор 890 образцов растительного материала в виде листочков и веточек в садах Алматинской, Жетысуйской и Абайской областях. Были разработаны праймеры для идентификации Erwnina amylovora и Venturia методом азотермической амплификации.
Была проведена валидация разработанных праймеров на положительных контролях коллекции лаборатории, а также на синтетических контролях. Нижний порог чувствительности разработанных праймеров составил 20-40 фг.
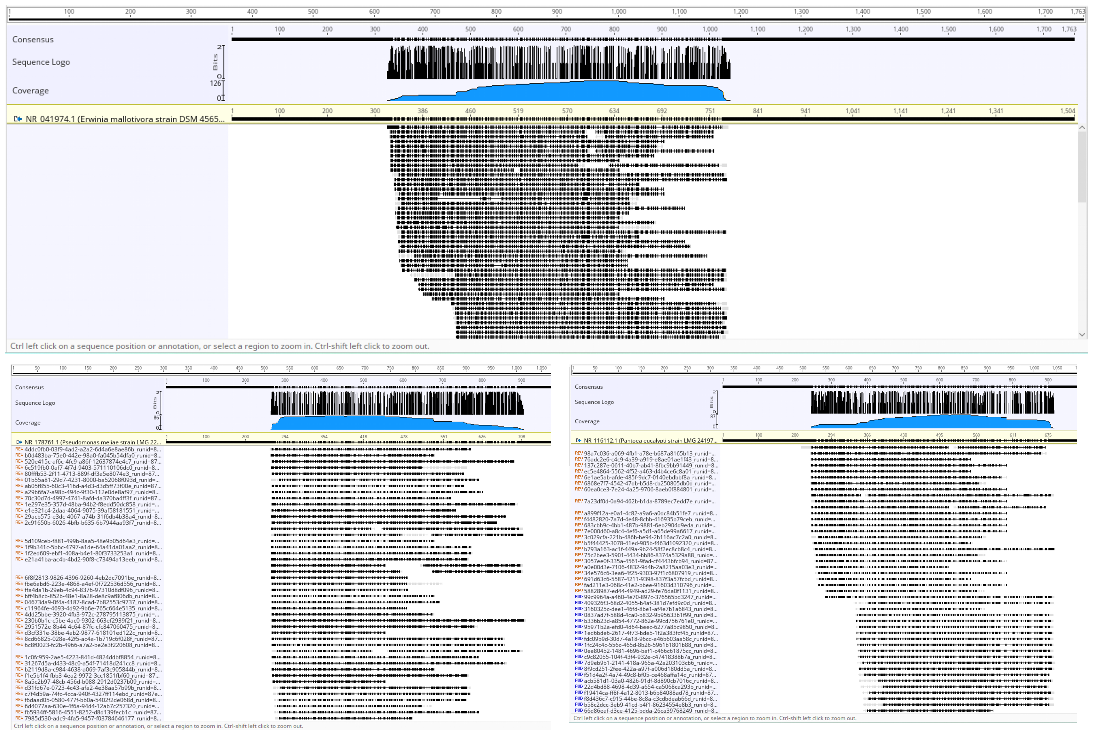
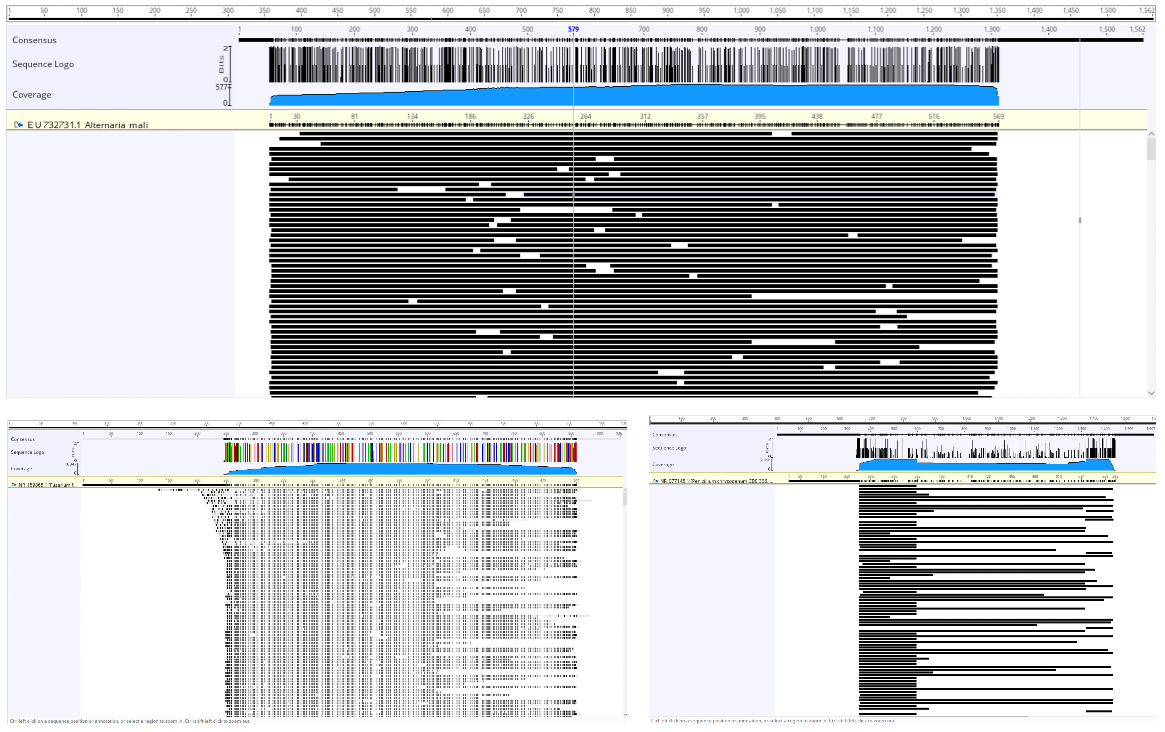
Был проведен метагеномный анализ для собранных образцов в садах и в диких популяциях. Метагеномный анализ проводился путем секвенирования генов 16sRNA, а также ITS1-ITS2 на платформе MinIon. Из патогенной микрофлоры, премущественно были выявлены бактерии рода Erwinia, эндофитная микрофлора была представлена Pseudomonas, Klebsiella и Pantoea. Сопутствующая микрофлора была представлена бактериями рода Pseudomonas. У растений в диких популяциях из эндофитной микрофлоры преимущественно была выявлена бактерия Pseudomonas fluorescens. Из патогенных грибков был выявлен род Alternaria, преимущественно вида Alternaria mali, вызывающий альтернариоз яблони. У боярышника и рябины среди патогенных грибков были выявлены Diplocarpon mespili, Alternaria и Monilinia johnsonii. Среди эндофитных грибков были выявлены следующие виды: Alternaria infectoria, Alternaria sclerotigenum, Aspergillus terreus, Penicillium chrysogenum, Fusarium lateritium. В диких популяциях были выявлены все вышеуказанные эндофитные виды грибков, в культурных садах только вид Alternaria infectoria.
В зависимости от исследуемого органа растения состав патогенной и эндофитной микрофлоры варьировал. В листьях были обнаружены: Pseudomonas, Klebsiella, Pantoea, Alternaria sclerotigenum, Aspergillus terreus. Alternaria mali. В то время как в веточках были выявлены: Erwinia, Pseudomonas fluorescens, Alternaria infectoria, Alternaria sclerotigenum, Aspergillus terreus, Penicillium chrysogenum, Fusarium lateritium.
Выявленные изоляты Erwnina amylovora были подтверждены также методом LAMP при использовании разработанных высокоспецифичных праймеров.
Достигнутые результаты за 2024 год
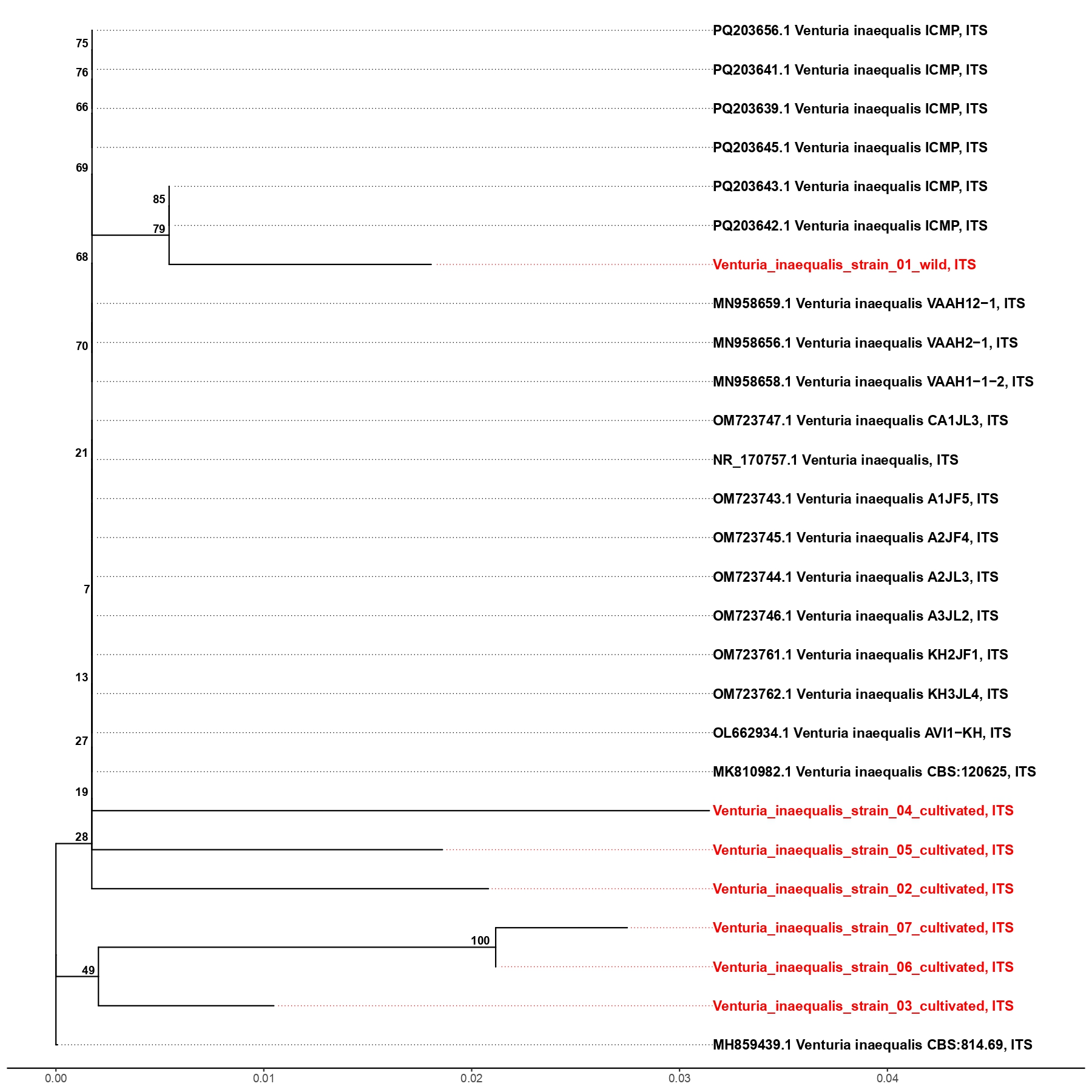
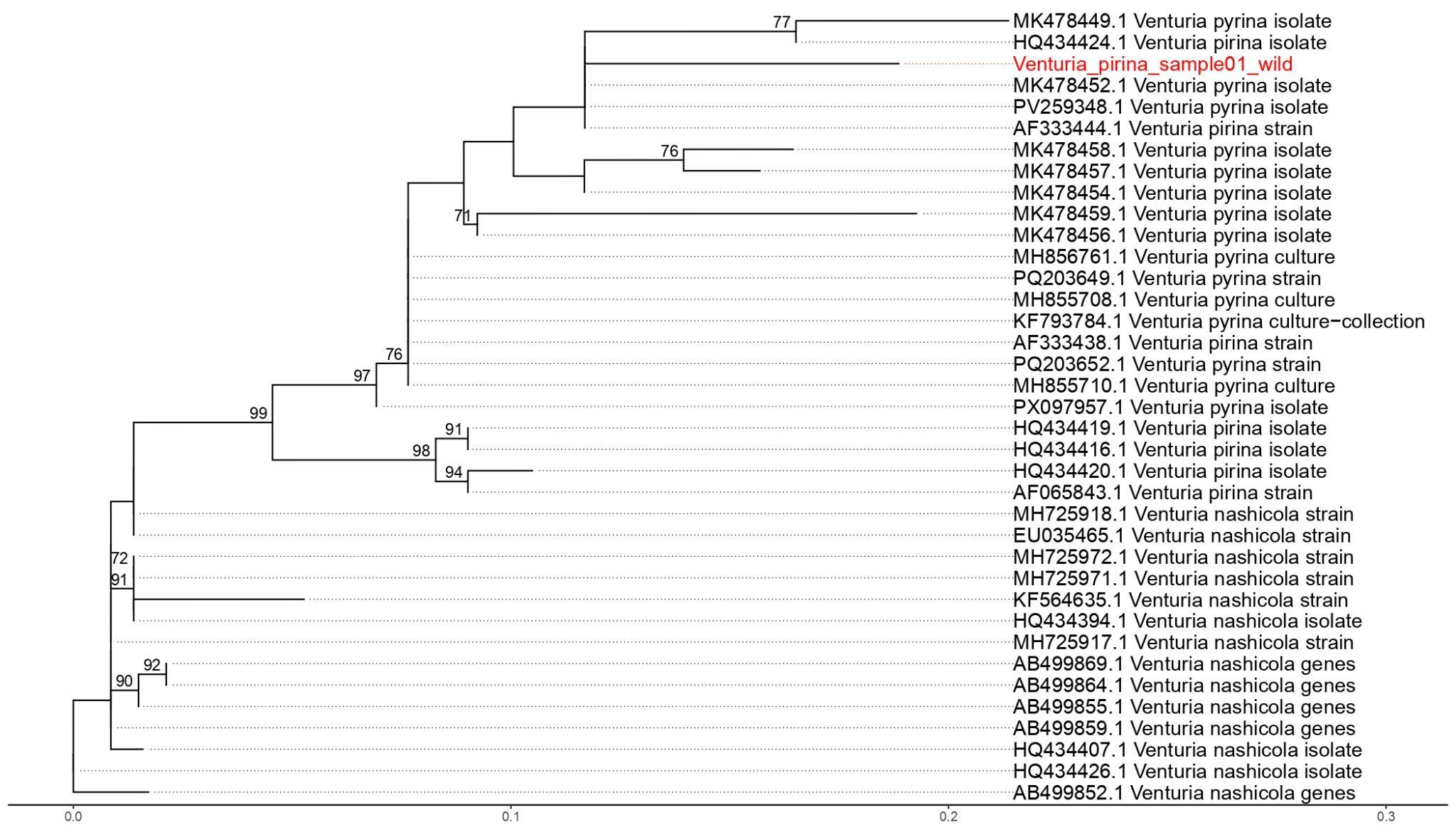
Было проведено полногеномное секвенирование 30 изолятов Erwnina amylovora и 8 изолятов Venturia. Наиболее генетическими близкими штаммами для Erwnina amylovora из базы данных NCBI являлись N434113.1 и FN666575.1. Большинство изолятов Erwnina amylovora относятся к R1 риботипу. Наибольшее количество вариаций было выявлено в плазмидном геноме и белок некодирующих регионах генома. Большинство вариаций, 54%, приходились на изоляты, выделенные у дикой яблони. Семь изолятов Venturia inaequalis показали наибольшее генетическое родство с расой 1, и один изолят с расой 2. Было выявлено 125 вариаций в 8 изолятах Venturia inaequalis, выделенных в географически отдаленных популяциях. Было выявлено 3 уникальные вариации в генах, кодирующих эффекторные белки у Venturia inaequalis раса 1. Было выялено 5 изолятов Erwnina amylovora, относящихся к А-генотипу при изучении CRR1 регионов генома. Было выявлено от 246 до 260 SNP среди 30 изучаемых изолятов. Изоляты Venturia inaequalis обладали гомологией, которая составилла 98,7%. Наибольшее генетическое расстояние составило 0.015, наименьшее генетическое расстояние составило 0,009. По результатам анализа Erwinia amylovora, по >3800 позиций генома, изоляты формируют три основных филогенетических кластера, различающихся в среднем по 147 SNP. Создана коллекция чистых культур Erwnina amylovora (30 изолятов) и Venturia inaequalis (8 изолятов).
Публикации и патенты
1. Kerimbek, N., Kapytina, A. I., & Taskuzhina, A. K., Gritsenko D.A. (2024). Aassessment of occurrence and diversity of apple viruses in south kazakhstan. Ġylym ža̋ne bìlìm, 1(2 (75)), 79-85.
Полученные результаты по задачам 2025:
Впервые в Казахстане проведен комплексный полногеномный и молекулярно-генетический анализ возбудителей бактериального ожога (Erwinia amylovora) и парши яблони (Venturia spp.) в популяциях дикой и культурной яблони, груши, рябины и боярышника. Сформирована коллекция из 1043 растительных образцов с GPS-метками, охватывающая ключевые регионы Казахстана и Кыргызстана. Проведено полногеномное секвенирование 7 изолятов E. amylovora и 8 изолятов V. inaequalis, что позволило выявить генетическое разнообразие, штаммы и расы патогенов, а также установить культуроспецифичные генетические паттерны и локальные очаги инфекции. Разработаны и валидированы LAMP-праймеры для быстрой диагностики, построены карты инфекционного фона. Результаты имеют значение для фитосанитарного мониторинга, диагностики и селекции устойчивых сортов плодовых культур.
Информация для потенциальных пользователей
Потенциал использования результатов проекта в научной и прикладной сфере очень высокий. Полученные данные о геномной вариабельности и вирулентности Erwinia amylovora и видов рода Venturia могут быть использованы для разработки высокоспецифичных систем детекции и мониторинга патогенов. Созданная референтная коллекция штаммов позволит лабораториям проводить диагностику и оценку патогенности изолятов, а разработанные подходы могут быть применены для предотвращения распространения инфекций в садах и диких популяциях плодовых культур.
Эффективность проведенных исследований подтверждается успешным выполнением всех запланированных этапов и достижением значимых научных и практических результатов. Впервые в Казахстане проведено полногеномное и молекулярно-генетическое изучение штаммов Erwinia amylovora и видов рода Venturia, циркулирующих в диких и культурных популяциях яблони и груши. Создана референтная коллекция штаммов и разработана информационная карта распространения патогенов, что обеспечивает научную основу для разработки эффективных методов диагностики и профилактики бактериального ожога и парши плодовых культур.
Публикации и патенты
- Pozharskiy, A.; Kostyukova, V.; Nizamdinova, G.; Gritsenko, D. Comparison of Different Methods of Molecular Detection of Erwinia amylovora in Plant Material. Curr. Issues Mol. Biol. 2025, 47, 1034. https://doi.org/10.3390/cimb47121034
- Kostyukova, V.; Pozharskiy, A.; Khusnitdinova, M.; Nizamdinova, G.; Gritsenko, D. Morphological and Molecular Characterization of Apple Scab (Venturia inaequalis) in Kazakhstan and Kyrgyzstan. Curr. Issues Mol. Biol. 2025, 47, 1011. https://doi.org/10.3390/
cimb47121011.
- Khusnitdinova, M., Taskuzhina, A., Kerimbek, N., Pozharskiy, A., Nizamdinova, G., Gritsenko, D. (2025). Analysis and Assessment of NDVI Variations in Response to Climate Change Among Wild Trees in the Zhongar Alatau Mountain Region. Geographic Approaches to Climate Change and Mitigation: Urban and Rural Perspectives (Volume 1). GCUE 2024. Advances in Science, Technology & Innovation. Springer, Cham. P. 105-113. https://doi.org/10.1007/978-3-031-92119-3_10.
- Khusnitdinova M., Taskuzhina A., Kerimbek N., Pozharskiy A., Nizamdinova G., Gritsenko D. BIODIVERSITY MAPPING OF MALUS SIEVERSII FORESTS IN SOUTHEASTERN KAZAKHSTAN USING GEOSPATIAL TOOLS // Proceeding of 3rd International Symposium on the Frontiers of Plant Diversity Conservation Research on the Pan-Third Pole Region (PDCR 2025). — Almaty. — August, 26-29 2025. — P.30.
- Yanin K., Taskuzhina A., Kerimbek N., Pozharskiy A., Nizamdinova G., Gritsenko D. TRANSCRIPTOMIC АNALYSIS OF MALUS SIEVERSII UNDER NATURAL CONDITIONS // Proceeding of 3rd International Symposium on the Frontiers of Plant Diversity Conservation Research on the Pan-Third Pole Region (PDCR 2025). — Almaty. — August, 26-29 2025. — P.26.
- Taskuzhina A., Kerimbek N., Makhambetov A., Mendybayeva A., Gritsenko D. COMPREHENSIVE ANALYSIS OF THE MICROBIAL COMMUNITIES AND GENOMIC DIVERSITY OF MALUS SIEVERSII IN KAZAKHSTAN // Proceeding of 3rd International Symposium on the Frontiers of Plant Diversity Conservation Research on the Pan-Third Pole Region (PDCR 2025). — Almaty. — August, 26-29 2025. — P.41.

А, Б, В — пораженные листья яблони ; Г — ветви с признаками бактериального ожога; Д, Е — плоды яблони пораженные паршой
Рисунок 1 — Симптомы поражения растений рода Malus фитопатогенами
Venturia inaequalis и Erwinia amylovora
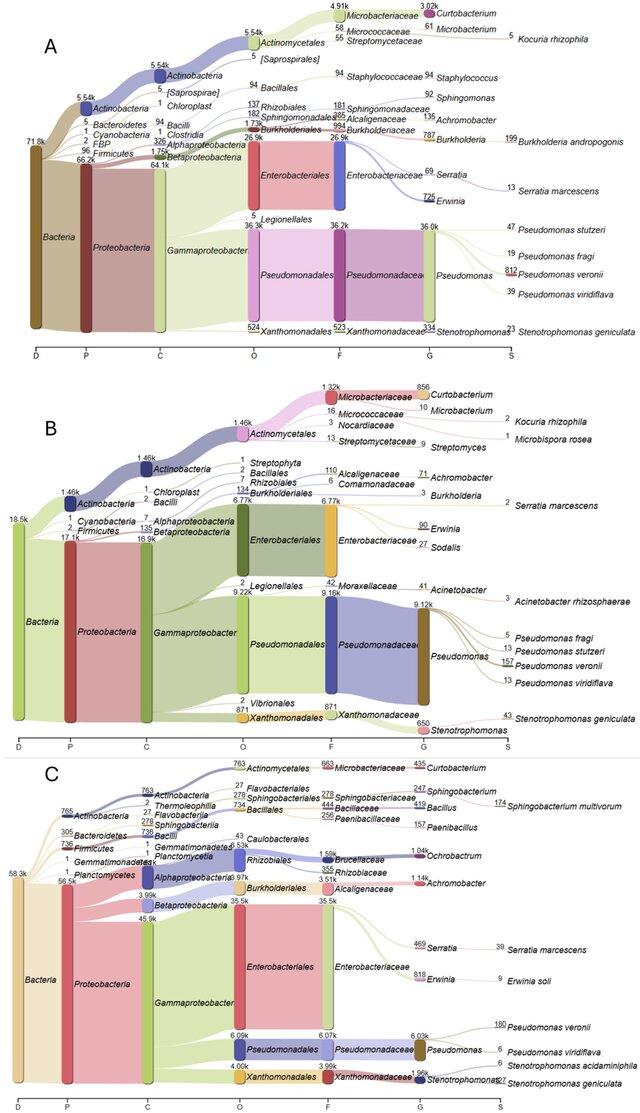
Рисунок 4 — Результаты секвенирования популяций из трех регионов.
(A) Регион Заилийский Алатау; (B) Регион Джунгарский Алатау; (C) Регион Кетпентау. Цвета обозначают различные таксономические группы
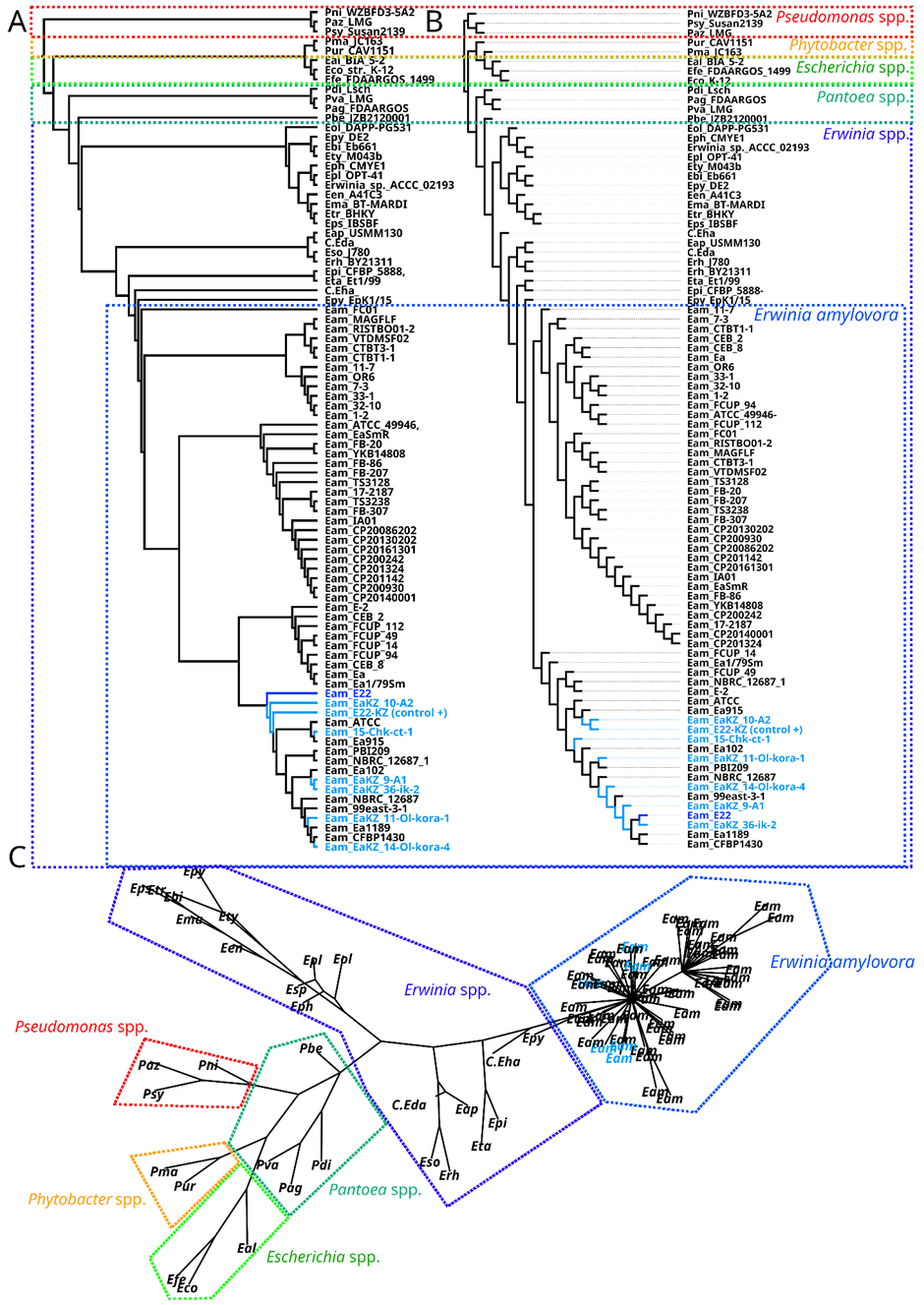
A) Филогенетическое дерево, построенное по конкатенированным генам; B) Консенсусное дерево, построенное на основе индивидуальных деревьев для генов rpsL, rplN, infB и hisS; C) Консенсусная сеть, построенная на основе индивидуальных деревьев для тех же генов Локальные изоляты показаны синим цветом
Рисунок 5 — Филогенетический анализ изолятов Erwinia amylovora
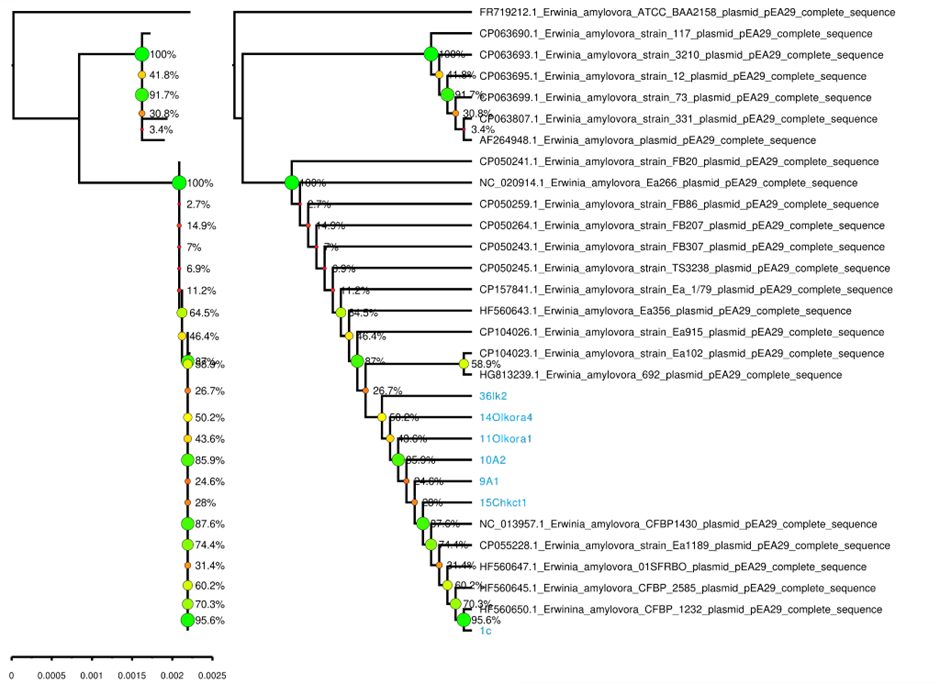
Рисунок 6 — Филогенетическое дерево по методу максимального правдоподобия на основе полных последовательностей плазмиды pEA29
Кладограмма справа демонстрирует топологию ветвления без учета расстояний
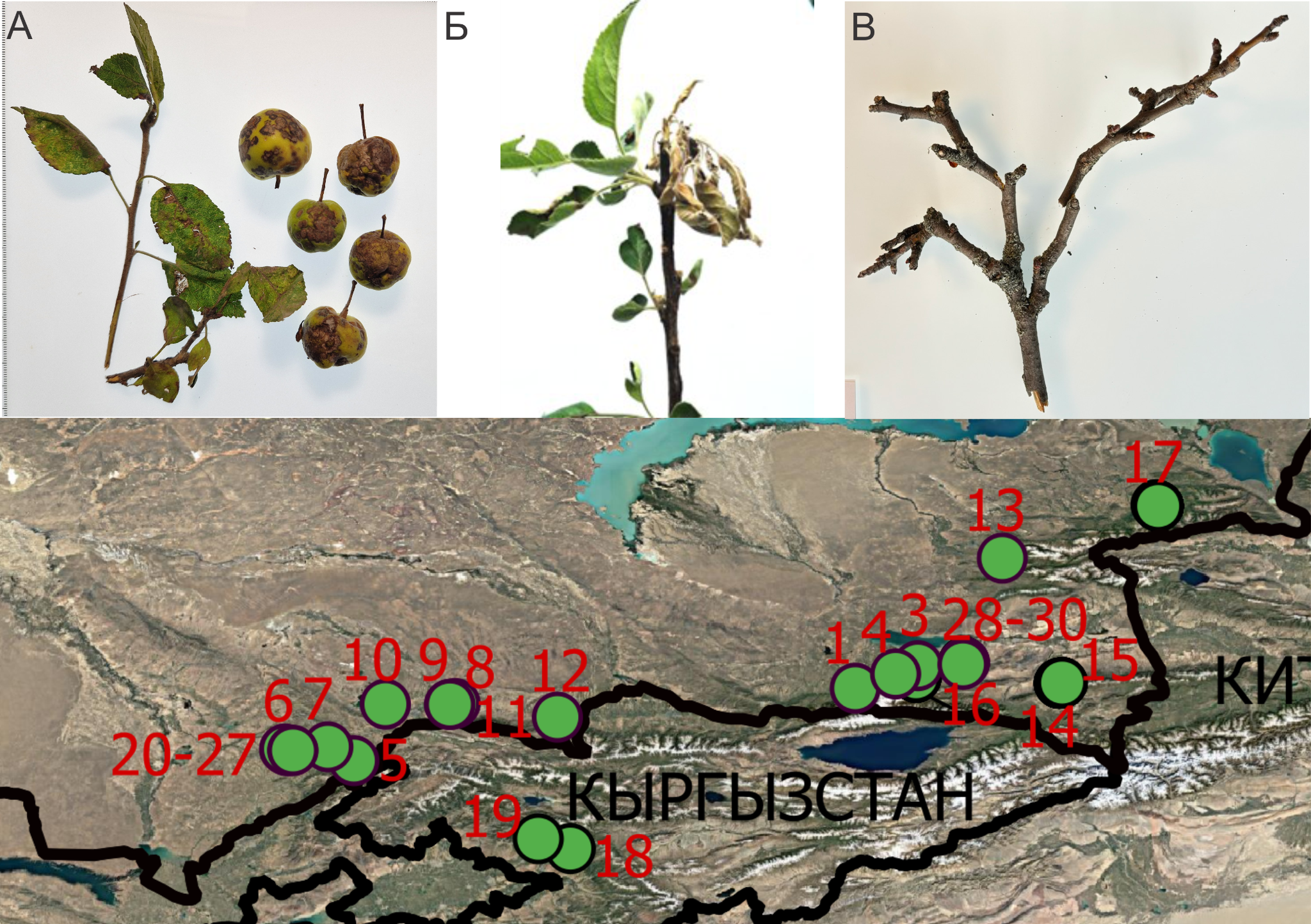
А — листья и плоды яблони с симптомами парши
Б и В — веточки яблони и груши с симптомами бактериального ожога
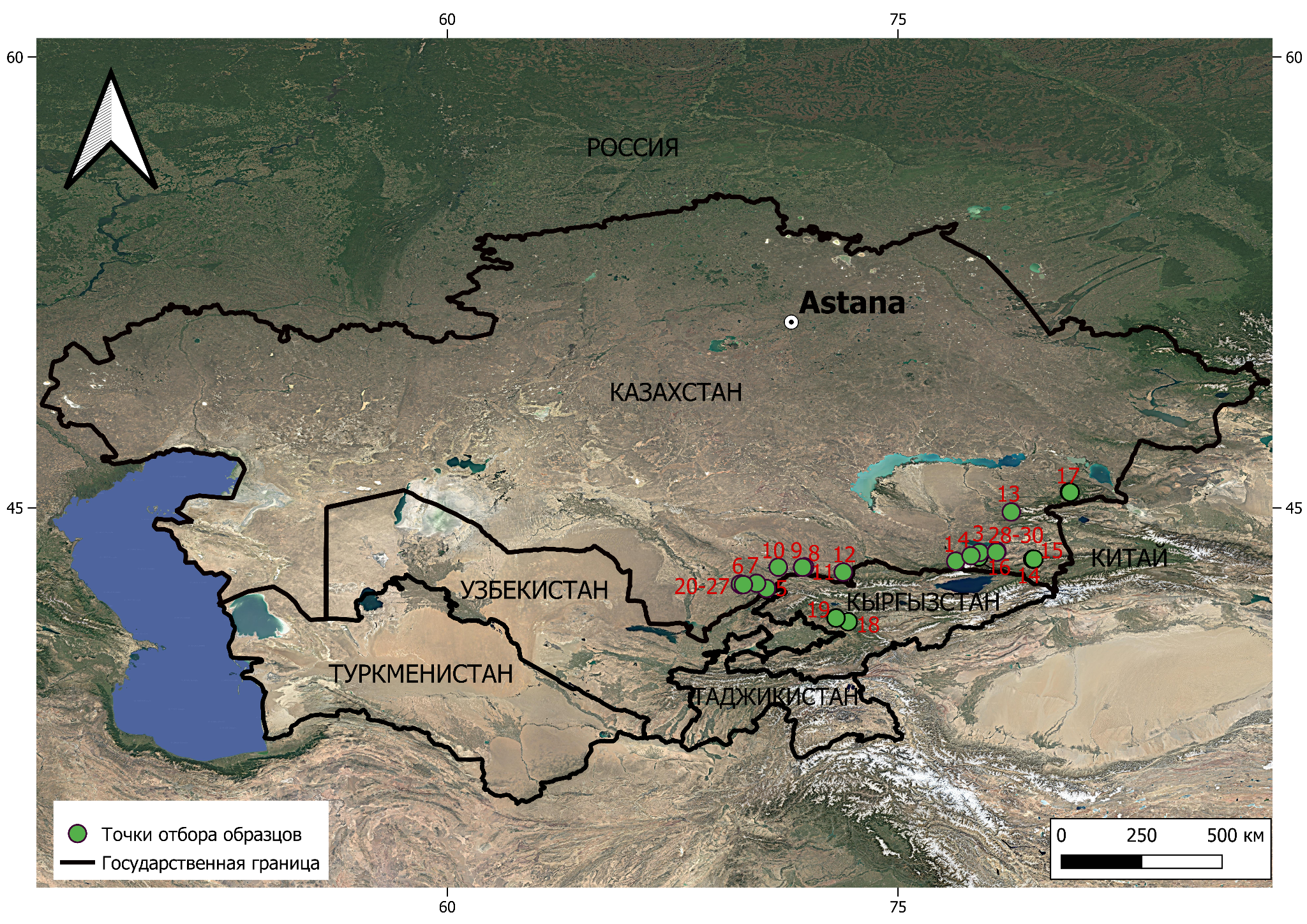
Рисунок 10 — Фрагмент карты территории отбора образцов
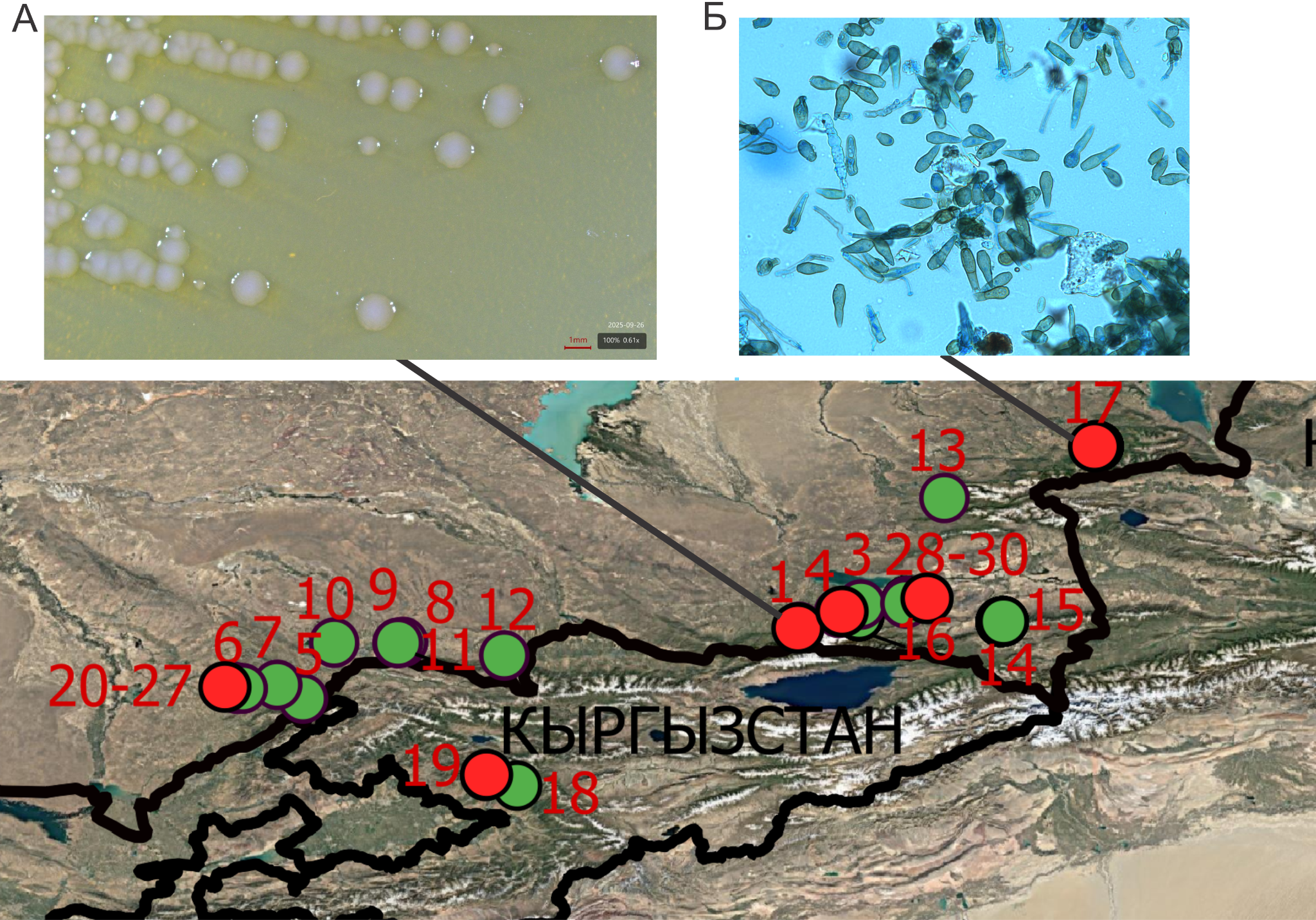
А — морфология колоний Erwinia amylovora с увеличением 0,61Х
Б — изображение результатов микроскопирования Venturia inaequalis с увеличением 40Х
Рисунок 11 — Фрагмент карты циркуляции Erwinia amylovora и Venturia inaequalis в популяциях дикой и культурной яблони и груши